数据的质量控制——fastQC
编者按
目前的高通量测序技术可以在单次运行中产生数亿个序列。在分析此序列以得出生物学结论之前,应该执行一些简单的质量控制检查,以获得较好的原始数据,并且确保数据中没有任何问题或偏差,本文就来介绍一款简单常用的质量检测工具fastQC。
大多数测序平台会生成一个QC报告作为其分析流程的一部分,但这通常只能识别由测序仪本身产生的问题。FastQC的开发和维护主要由Babraham Bioinformatics实验室负责,旨在提供一个可以发现来自测序平台或起始文库的问题的QC报告。
FastQC可以以两种模式运行。它可以作为独立的交互式应用程序运行,用于临时分析少量的FastQ文件,也可以以非交互模式运行,用于集成到较大的分析流程中,用于系统地处理大量文件。
1 下载和安装
FastQC是用java写的,所以需要一个Java Runtime Environment,还需要Picard BAM/SAM库,软件支持多个系统平台,包括windows版、Linux和MacOS。本文使用Linux环境。
FastQC官网下载:http://www.bioinformatics.babraham.ac.uk/projects/fastqc/
这里我们选择下载编译好的程序,上传软件安装包到Linux服务器,使用unzip命令来进行解压缩。然后进入解压缩文件,fastqc文件即是主程序,使用chmod u+x 命令修改为可执行权限。
1 使用的命令
我们在服务器上用命令行来运行fastQC:
fastqc[-o output dir] [--(no)extract] [-f fastq|bam|sam] [-c contaminant file]seqfile1 .. seqfileN
-o用来指定输出文件的所在目录,生成的报告的文件名是根据输入来定的,注意是不能自动新建目录的。输出的结果是.zip文件,默认自动解压缩,命令里加上--noextract则不解压缩。
-f 用来强制指定输入文件格式,默认自动检测。
-c 污染物选项,输入的是一个文件,格式是Name[Tab] Sequence,#开头的行是注释,里面是可能的污染序列,如果有这个选项,FastQC会在计算时候评估污染的情况,并在统计的时候进行分析。
-q 会进入沉默模式,指定这个选项的时候,程序不会实时报告运行的状况,即不出现下面的提示:
Startedanalysis of target.fq
Approx5% complete for target.fq
Approx10% complete for target.fq
1 fastQC报告解读
打开生成的HTML格式的结果报告,如下图所示:
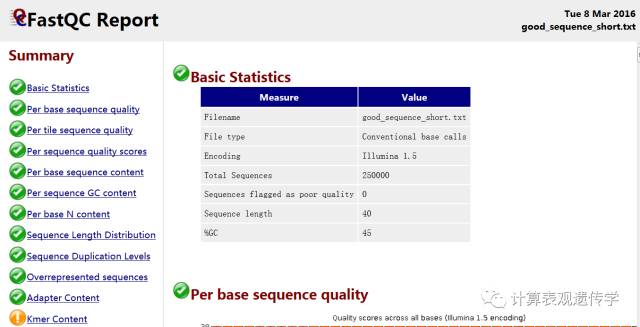
Summary 概要
本部分就是整个报告的目录,整个报告分成若干个部分。合格会有个绿色的对勾,警告是黄色叹号,不合格是红叉。
Basic Statistics 基本信息
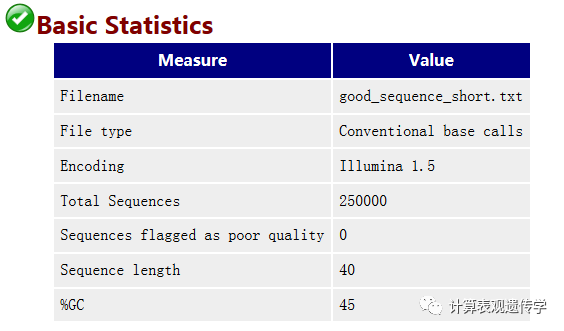
Encoding指测序平台的版本和相应的编码版本号,可推测是Phred33 或是Phred 64 质量分数的编码方式。
Total Sequences输入文本的reads的数量。
Sequence length 测序的长度
%GC 是我们需要重点关注的一个指标,这个值表示的是全部序列中的GC含量,这个数值一般是物种特意的,比如人类细胞就是42%左右。
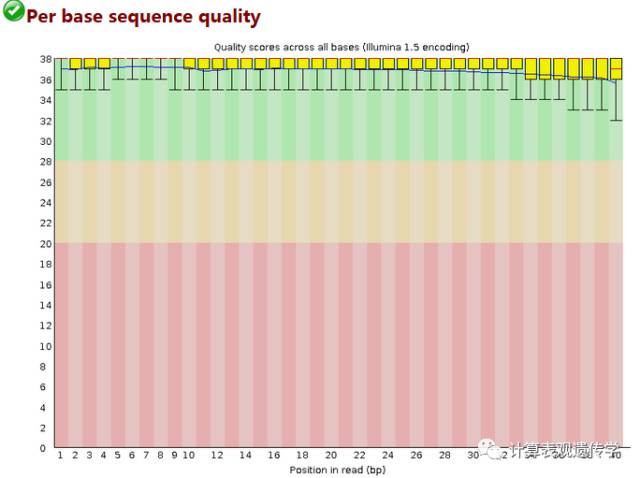
用箱式图的方式展示数据质量,图中每1个位置,都是该位置的所有序列的测序质量的一个统计。纵轴是质量得分,Q =-10*log10(p),p为测错的概率。所以一条reads某位置出错概率0.01时,其quality就是20。横轴是测序序列的位置。蓝色线是各个位置的平均值的连线。一般要求此图中,所有位置的10%分位数大于20,也就是常说的Q20过滤。
所以上面的这个测序结果质量很好。如果任何碱基质量低于10,或者是任何中位数低于25报警,如果任何碱基质量低于5,或者是任何中位数低于20报错。
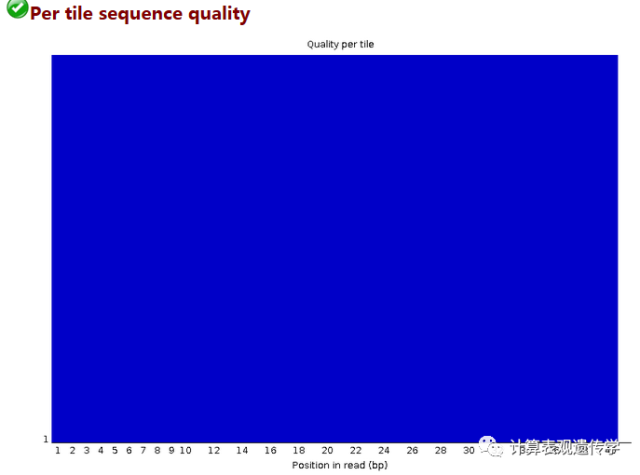
这一模块是检查在测序平台上,reads中每一个碱基位置在不同的测序小孔之间的偏离度,偏离度越高,碱基质量越差。纵轴表示测序小孔,蓝色表示低于平均偏离度,越红则说明偏离平均质量方差越多,也就是说质量越差,本图中都是蓝色表明质量很好。如果出现质量问题可能是短暂的,如有气泡产生,也可能是长期的,如在某一小孔中存在杂质。偏离度小于平均值2以上报警,偏离度小于平均值5以上不合格。
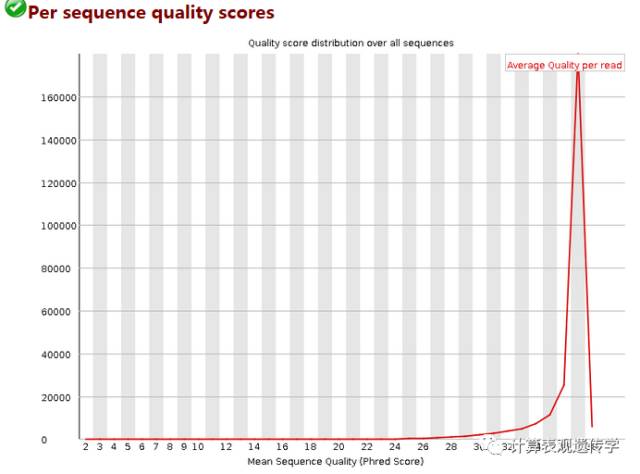
这是为了检测一部分质量特别差的reads,如果有则会在图上出现多个峰,如在测序仪边缘的reads。纵轴是reads数目,横轴是质量分数,代表不同Phred值对应了多少的reads。
本图中,测序结果主要集中在高分中,证明测序质量良好。当峰值小于27(错误率0.2%)时警报,当峰值小于20(错误率1%)时不合格。
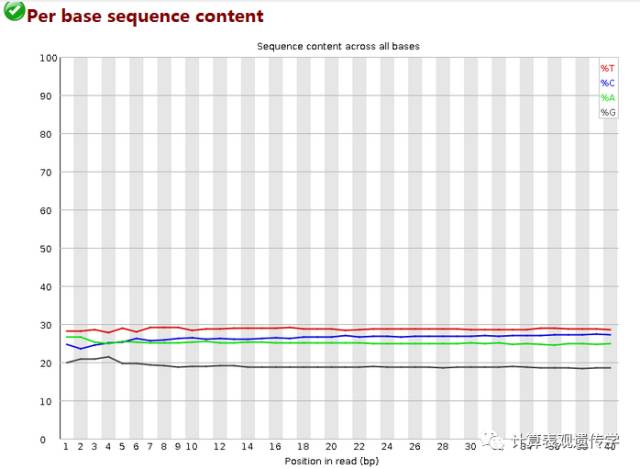
展示碱基含量分布,它根据碱基的位置对每个位置上的A,C,G,T的含量进行统计,横轴为位置,纵轴为百分比。正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该平行且接近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresented sequence的污染。当所有位置的碱基比例一致的表现出bias时,即四条线平行但分开,往往代表文库有bias (建库过程或本身特点),或者是测序中的系统误差。当任一位置的A/T比例与G/C比例相差超过10%发出警报,超过20%则数据不合格。
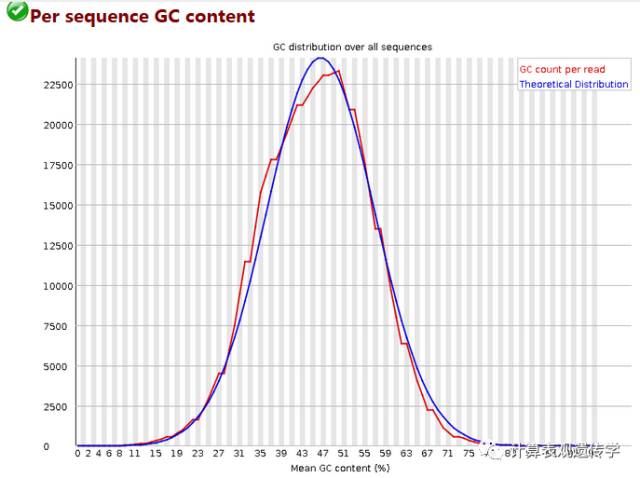
图中红色曲线是实际的测序GC含量分布图,而蓝色曲线则是理论分布(正态分布,不过均值不一定都是50%,而是由平均GC含量推断的)。如果红色曲线形状存在比较大的偏差,往往是由于文库的污染造成的。红色曲线越平滑越好,越接近蓝色曲线越好。形状接近正态但偏离理论分布的情况提示我们可能有系统偏差。偏离理论分布的reads超过15%时发出警报,超过30%时报不合格。
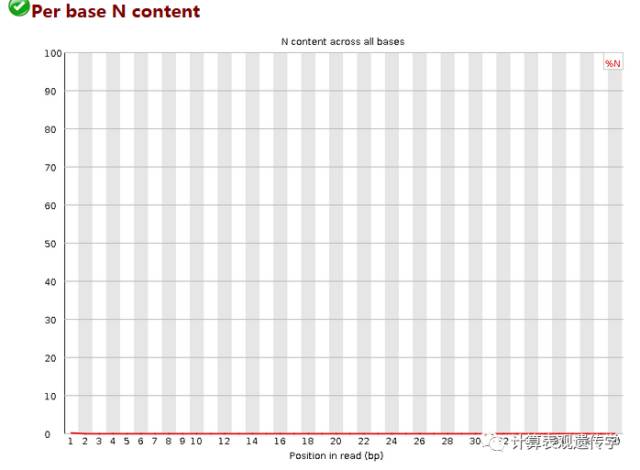
纵轴是百分含量,横轴是read的位置,当测序仪不能确切地测定出某一个碱基时就会标注为N,正常情况下N的比例是很小的,所以图上常常看到一条直线。当看到有峰时,说明测序出了问题。当任意位置的N的比例超过5%警报超过20%不合格。
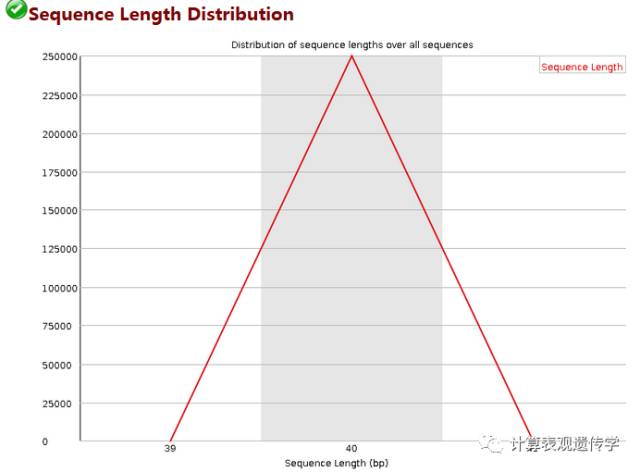
每次测序仪测出来的长度在理论上应该是完全相等的,但是总会有一些偏差,如此图中,40bp是主要的,但是还是有少量的39和41bp的长度,不过数量比较少,不影响后续分析,当测序的长度有很大不同时,则表明测序仪在此次测序过程中产生的数据不可信,但对于某些测序平台,具有不同的read长度是完全正常的。当reads长度不一致时警告,当有长度为0的read时不合格。
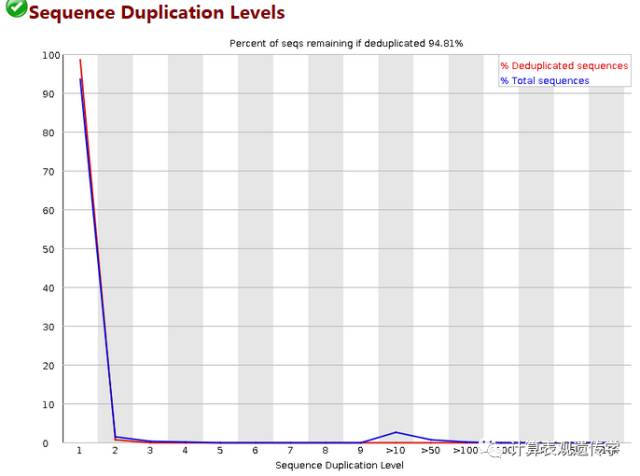
横轴为reads重复的次数,纵轴为重复次数对应的reads占不重复的reads的比例。测序深度越高,越容易产生一定程度的duplication,这是正常的现象,但如果duplication的程度很高,那么表明存在富集的偏好(enrichment bias)(比如:测序过程中的PCR重复,转录组测序中某些基因表达量高),序列重复比例越高,则表明实际有用的序列越少。图中有蓝红两条线,蓝色线表示的是文件中所有的序列中duplicate程度的分布,红色线表示的是去冗余之后的序列,含量表示的在全部序列都考虑时不同冗余程度的序列所占的比例。重复reads占总数的比例大于20%时警报,大于50%时不合格。
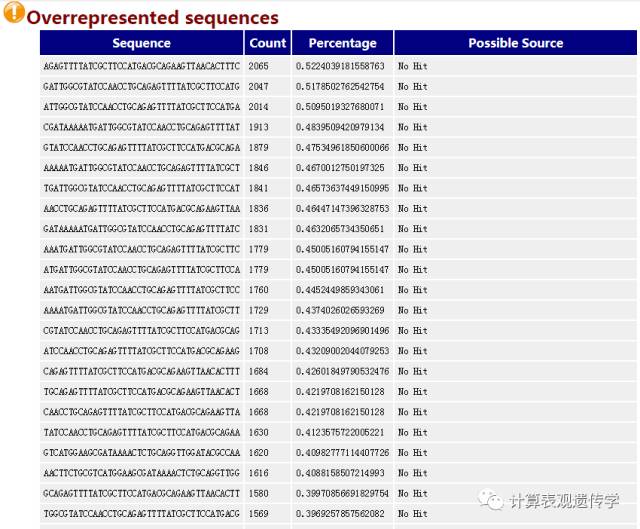
如果有某个序列大量出现,就叫做over-represented。标准是占全部reads的0.1%以上。但是因为用的是Duplicate sequences前200,000条数据,所以有可能over-represented reads不在里面,参考意义不大。
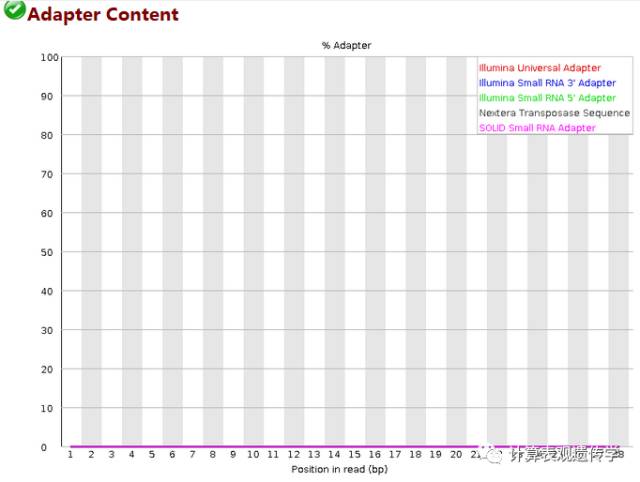
此图衡量的是序列中两端adapter的情况,如果在fastqc分析的时候-a(指定含adapters序列文件)选项没有内容,则默认使用图例中的通用adapter序列进行统计。含有adapter超过所有reads的5%的警告,超过10%不合格。
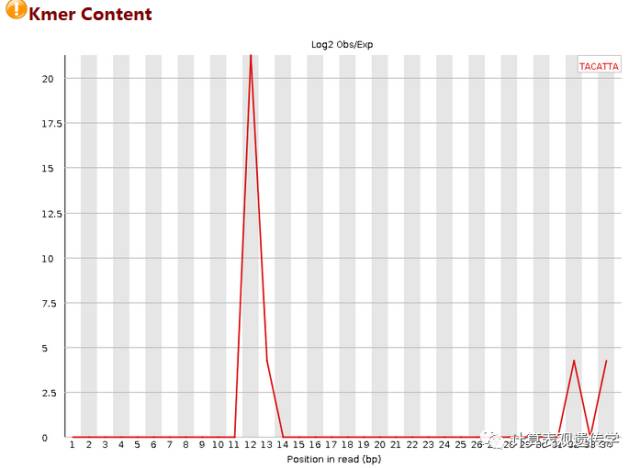
这个图统计的是,在序列中某些特征的短序列重复出现的次数,我们可以看到11-14bp的时候图例中的短序列出现了非常多的次数,一般来说,出现这种情况,要么是adapter没有去除干净,而又没有使用-a参数;要么就是序列本身可能重复度比较高,如建库PCR的时候出现了bias。
更多详细信息参考fastQC使用说明:http://www.bioinformatics.babraham.ac.uk/projects/fastqc/
©著作权归作者所有:来自51CTO博客作者mob604756f4ef89的原创作品,如需转载,请注明出处,否则将追究法律责任